Die Bioinformatik ist eine recht junge Teildisziplin der Informatik, die sich vorwiegend mit der Auswertung biologischer Daten, sowie deren Speicherung und Strukturierung beschäftigt. Zu Beginn waren es meist die DNA-Sequenzen von Genen bzw. Genomen, die in großen Datenbanken gespeichert und analysiert wurden. Inzwischen gehören zur Bioinformatik u.a. aber auch die Vorhersage von Protein- und RNA-Strukturen, Erstellung von phylogenetischen Bäumen und die Untersuchung von Wechselwirkungen zwischen Substanzen in der Pharmazie.
Deshalb spielt die Bioinformatik inzwischen auch in den traditionellen Naturwissenschaften wie z.B. der Molekularbiologie, Biochemie, Pharmazie und Genetik eine immmer größer werdende Rolle.
Wichtige Schwerpunkte der Bioinformatik sind:
Analyse von DNA Sequenzen / Phylogenien

Mit der Erfindung von Methoden zur Bestimmung von Nukleotidsequenzen und durch die Bestrebungen der verschiedenen Genomprojekte füllten sich die Datenbestände der Biologen mit immer mehr Gensequenzen der unterschiedlichsten Organismen. Eine der ersten Anwendungen in der Bioinformatik war demnach die Suche nach bestimmten Sequenzen in den Gendatenbanken. Mittlerweile gibt es eine Fülle an Algorithmen und Werkzeugen, um in den riesigen Sequenzdatenbanken mit akzeptabler Performance nach Sequenzmustern zu suchen.
Ein weiteres Gebiet der Bioinformatik ist die sog. Phylogenie-Rekonstruktion. Dabei werden die Verwandtschaftsverhältnisse von Organismen bzw. Arten an der Abweichung ihrer DNA-Sequenzen gemessen. Die daraus entstehenden Abstammungsbäume zeigen die evolutionäre Distanz der Organismen auf. Grundlage sind hier Algorithmen die den Unterschied zwischen zwei oder mehreren Sequenzen berechnen und ein Alignment, also eine möglichst optimale Anordnung der Sequenzen liefern.
Strukturvorhersage von Proteinen

Nach der Entschlüsselung des genetischen Codes und der Sequenzierung vieler Gene bzw. ganzer Genome ist es möglich die Aminosäuresequenz der verschiedenen Genprodukte, also von Proteinen anzugeben. Diese aminosäureketten fallten sich in einem komplizierten Verfahren zum fertigen Protein. Die bloße Aminosäuresequenz sagt also noch nicht viel über die dreidimensionale Struktur aus. Das Wissen darüber ist aber entscheidend für das Verständnis verschiedener Merkmale des Proteins, wie etwa die enzymatische Aktivität. Daher ist die Vorhersage der räumlichen Struktur ausgehend von der bekannten Sequenz eine der zentralen und schwierigsten Aufgabenstellungen in der Bioinformatik.
Eine heute am meisten verwendete Methode beruht auf dem sog. Homology Modelling. Um die bisher unbekannte Struktur eines neuen Proteins zu finden, sucht man nach einem möglichst homologen, also von der Sequenz her ähnlichen Protein, dessen Struktur bekannt ist. Dieses benutzt man als Vorlage um ein Modell des Zielproteins zu erstellen.
Vorhersage von RNA Sekundärstrukturen
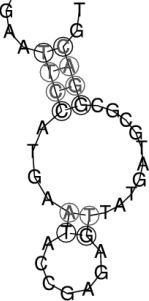
In der Vergangenheit wurde die RNA hauptsächlich als Informationsträger von der DNA im Zellkern zu den Ribosomen im Zellplasma angesehen. Erst mit der Entdeckung mehrerer verschiedener RNA-Typen und der RNA-Welt-Hypothese wuchs ihre Bedeutung für viele Funktionen in der Zelle. Im Gegensatz zur DNA kann die einzelsträngige RNA selbst katalytisch aktiv werden.
Um katalytische Zentren in einem RNA-Molekül zu finden, ist es wichtig ihre Sekundär- bzw. Tertiärstruktur zu kennen. Im Gegensatz zu Proteinen ist es bei RNA leichter die Sekundärstruktur allein anhand der Nukleotidsequenz zu berechnen, z.B. mit dem Vienna RNA Package. Damit wird die Struktur anhand von Minimum Free Energy Structures sowie der Basenpaarungswahrscheinlichkeiten berechnet.
Biologische Datenbanken
Eine Hauptaufgabe der Bioinformatik ist es Methoden zur Speicherung, Integration und Analyse von biologischen Daten aller Art bereitszustellen. Inzwischen gibt es große im Internet zugängliche Datenbanken für verschiedenste Typen von Genom-, Protein- und Stoffwechselinformationen. Eine entscheidende Komponente ist dabei die Quervernetzung und Annotierung der verschiedenen Informationen in den vielen Datenquellen. Außerdem müssen unterschiedlichste Datenarten integriert werden, etwa experimentelle Rohdaten, aufbereitete Experimentdaten und textuelle Kommentare. Zuletzt sind natürlich effektive Suchmethoden erforderlich, um in den vielen Datenquellen die gesuchten Informationen zu finden.
Genchips
Genchips bzw. DNA-Microarrays sind eine recht junge Methode um die unterschiedliche Genexpression in Zellen verschiedener Gewebe bzw. unter verschiedenen Einflüssen zu charakterisieren. Das Wissen wann welche Gene in der Zelle exprimiert werden, hilft u.a. beim Verständnis von metabolischen Netzwerken und der Herstellung von Arzneimitteln.
Dieses Gebiet beschäftigt sich mit den Techniken der mathematischen Modellierung und Computersimulation biologischer Systeme. Ein Hauptziel hierbei besteht in der Untersuchung physiologische Körperfunktionen und pathologische Vorgänge um beispielsweise individuelle Krankheitsverläufe oder die Reaktion des Körpers auf die Gabe von Medikamenten vorherzusagen. Ein weiteres Ziel theoretischer Modellbildung ist Erarbeitung und Validierung neuer biologischer Konzepte. Die Modellierung bildet somit eine Brücke zwischen experimenteller Biologie / klinisch angewandter Medizin auf der einen und mathematischer Theoriebildung / Methoden der Computersimulation auf der anderen Seite.


